La biodiversité est constituée par la diversité des organismes vivants. Les chercheuses et chercheurs en biologie étudient par exemple les espèces en les identifiant, en les comparant, en les dénombrant. En s’intéressant à une espèce particulière, les scientifiques du vivant peuvent aussi identifier, comparer, et dénombrer les formes génétiques différentes qui la constituent. La discipline de la génétique des populations analyse ainsi les variations génétiques à l’intérieur d’une population et entre les populations d’une même espèce, dans le cadre de la biologie évolutive. Elle permet de mieux comprendre l’histoire évolutive des espèces, mais aussi de quantifier la modification de la diversité génétique dans les populations. Pour ce faire, elle s’appuie sur des marqueurs génétiques, c’est-à-dire des éléments qui se transmettent d’une génération à l’autre, comme par exemple, chez un individu, certains caractères de forme, de couleur ou de propriété physiologique ou bien plus récemment des marqueurs directement localisés sur l’ADN1. Les différentes formes d’un même marqueur servent à distinguer les individus d’une même population, ce qui permet de quantifier la diversité génétique.
Dans les années 1860, Gregor Mendel, abbé autrichien de Brno, alors qu’il manipulait et croisait dans le jardin de son monastère des plants de pois (Pisum sativum), observa que la couleur des graines (verte ou jaune) et leur forme (ridée ou lisse) possédaient une hérédité simple. Ces deux marqueurs génétiques initiaux, de type morphologique, ont été les premiers historiquement observés en biologie. Il relata cette observation dans un article qui marque la découverte des lois de l’hérédité, à la base de la génétique 2.
Quel est le sens de cette soif de données pour notre pratique scientifique ? Ces données sont-elles en mesure d’aider à répondre à la disparition des vivants que nous étudions ?
Or, depuis le milieu du vingtième siècle, la diversité génétique d’un nombre toujours croissant d’espèces a été étudiée à l’aide d’un type particulier de marqueurs génétiques : les marqueurs moléculaires3. Obtenus à partir de l’ADN extrait des individus, ces marqueurs révèlent directement la variabilité de l’endroit du génome analysé. La communauté scientifique considère généralement aujourd’hui que ces techniques ont grandement amélioré notre connaissance de la biodiversité et de son évolution. Cependant, depuis deux décennies, nous constatons une course aux données passant par la multiplication des marqueurs, obtenus grâce à des méthodes de plus en plus complexes, requérant des instruments spécialisés (séquenceurs), des produits chimiques multiples et une consommation d’énergie accrue : l’ADN se conserve idéalement dans des congélateurs à ‑80°C et les données sont analysées grâce à de multiples serveurs informatiques. De plus, l’échantillonnage de certaines espèces à travers les continents implique des déplacements nombreux : l’empreinte écologique de l’ensemble de la démarche est à considérer.
Quel est le sens de cette soif de données pour notre pratique scientifique ? Ces données sont-elles en mesure d’aider à répondre à la disparition des vivants que nous étudions ? Devons-nous vraiment mesurer toujours plus, pour augmenter la précision de nos observations ? Et donc, par voie de conséquence, augmenter encore l’empreinte écologique de nos métiers ?
Nous nous interrogeons ici sur l’usage, depuis 30 ans, des marqueurs génétiques. Nous sommes toutes deux chercheuses en génétique des populations et nous nous questionnons sur le sens de la pratique de notre discipline de recherche. Confrontées à de continuelles avalanches de données, aux promesses techniques sans cesse renouvelées et à leur influence sur le financement de nos recherches, nous sommes conduites à questionner la prééminence de la technologie et ses conséquences environnementales, pour repenser le sens de notre discipline.
Une revue indicatrice des évolutions de notre discipline
Afin de bien saisir l’ampleur de l’usage des marqueurs moléculaires ces trente dernières années, regardons le cas de la revue Molecular Ecology. Créée en 1992, celle-ci est devenue une revue internationale incontournable pour publier des travaux de génétique des populations — elle reflète donc les évolutions de notre discipline ces trois dernières décennies.
Pour visualiser l’évolution du nombre de marqueurs mobilisés par article au cours du temps, nous avons analysé deux articles, choisis au hasard, portant respectivement sur une espèce végétale et une espèce animale, chaque année, depuis la première parution en mai 1992.
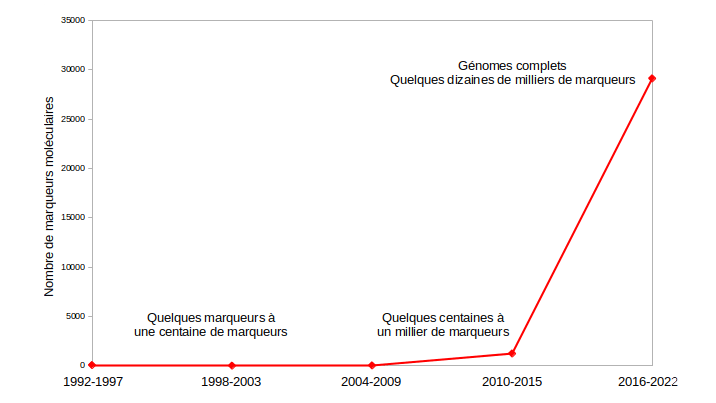
Ce tour d’horizon montre que si, pendant deux décennies, le nombre de marqueurs par étude était limité à moins d’une centaine, il atteint désormais plusieurs milliers, voire plusieurs centaines de milliers (figure 1). Cette évolution est inhérente aux techniques utilisées : le séquençage4 des génomes5 génère beaucoup plus de données (en nombre de mutations ponctuelles de l’ADN ou SNP6 ) que les autres marqueurs. Le grand développement du séquençage de l’ADN s’accompagne de l’augmentation toujours plus grande du nombre de marqueurs, ainsi que du nombre d’espèces pouvant être décrites. Nos propres travaux en génétique des populations s’inscrivent dans cette évolution. Nous étions préoccupées dans nos approches théoriques, publiées au tournant du siècle dans la revue Molecular Ecology, par la question du choix du type de marqueur ou du nombre de marqueurs à utiliser 7. Avec les espèces d’arbres que nous avons étudiées, nous sommes passées de l’utilisation de quelques marqueurs à l’étude de leurs génomes complets.
L’obsolescence des techniques plus anciennes trouve en principe une justification : la très grande accumulation de données est censée fournir des connaissances de plus en plus fines sur l’état et l’évolution temporelle de la biodiversité et pour un nombre d’espèces de plus en plus grand. Et cette désuétude s’accompagne également d’une obsolescence des outils : dans notre parcours, nous avons assisté, pour les études de génétique des populations, à la succession de plusieurs générations de machines, de serveurs et d’algorithmes, de complexité croissante. Les équipements sont ainsi abandonnés et, dans certains cas, certaines expériences ne peuvent parfois plus être réalisées de ce fait. Par exemple, dans notre propre laboratoire, nous ne pouvons étudier certains marqueurs très populaires qu’en les séquençant, alors qu’il était possible de les étudier plus simplement durant les dernières décennies, en tout cas sans mobiliser la bio-informatique. Nous contribuons ainsi à une éthique collective du gaspillage8.

Croyances et promesses techniques
Cette accumulation de techniques et de données de séquençage est portée par des discours scientifiques qui mettent souvent d’abord en avant la technologie et la méthodologie avant la question scientifique. Comme elle se décrit elle-même, la revue Molecular Ecology « publie des articles qui utilisent des techniques de génétique moléculaire pour répondre à des questions d’écologie, d’évolution, de comportement et de conservation9 ». Étant donné la prolifération et l’hyperspécialisation des revues académiques, Molecular Ecology n’a pas vocation à couvrir tout le champ de l’écologie. Elle se positionne à la confluence de l’écologie et des sciences de l’évolution. Mais, comme l’indique son nom — « écologie moléculaire » — les questions scientifiques autour de l’écologie y sont entourées par les outils techniques. Ces questions apparaissent en second dans la description donnée par la revue, tendant à mettre en avant une technoscience, déterminée par la technique et non par le questionnement scientifique.
Nos pratiques sont imprégnées d’un véritable « technosolutionnisme », plaçant cette foi dans le potentiel des technologies à résoudre les problèmes écologiques et en leur attribuant pour cela des qualités quasiment mystiques et magiques.
Plus généralement, notre communauté scientifique est en prise avec une véritable « économie de la promesse », aux accents technophiles, voire « techno-théologiques » selon l’expression de Bernadette Bensaude-Vincent10. En effet, les promesses techno-scientifiques s’engagent à résoudre les grands problèmes de société et notamment d’écologie, sous forme de discours, de moyens financiers et matériels, de production de connaissances, de construction de nouveaux réseaux sociotechniques11.
Il ne s’agit pas seulement de la croyance en la neutralité de la technologie, qui confine parfois au mythe12. Il existe aussi une foi quasi aveugle dans la technologie considérée comme une panacée. Nos pratiques sont imprégnées d’un véritable « technosolutionnisme13 », plaçant cette foi dans le potentiel des technologies à résoudre les problèmes écologiques et en leur attribuant pour cela des qualités quasiment mystiques et magiques14.
À lire sur Terrestres Vincent Devictor, « Anthropocène, dis-moi, combien tu t’appelles ? », mai 2019
Dans notre cas particulier, cette économie de la promesse fonctionne surtout dans le champ des applications de la génétique — ou génomique15 — des populations à la conservation des espèces, prises individuellement, ou bien à l’adaptation de leurs populations au réchauffement climatique. Nos travaux sont en effet produits dans le cadre de projets financés par différentes institutions : le Bureau des Ressources Génétiques (BRG) devenu Institut Français de la Biodiversité puis Fondation pour la Recherche sur la Biodiversité (FRB), ainsi que l’Agence Nationale de la Recherche (ANR), les Régions, l’Europe, ou des fondations privées. Quelles paroles les financements de ces recherches portent-ils ?
« Je peux le volatiliser et l’éterniser en le réduisant à un nombre, à un pur rapport numéral »
Martin Buber
Un examen de quelques projets financés ces dernières années par l’Agence Nationale de la Recherche et contenant les mots clés « génétique » et « population » permet d’illustrer la nature de ces promesses, pour toute une gamme de modèles biologiques. Il est ainsi question entre autres d’« améliorer la durabilité de l’élevage » grâce aux séquençages de populations expérimentales, d’utiliser la génomique des populations pour l’adaptation des fruitiers cultivés, d’explorer largement la diversité génétique pour une « production durable » de « réinvestir la diversité allélique de la betterave par le développement de nouveaux outils -omics et de nouvelles stratégies de sélection16 ».
La menace prégnante des changements climatiques redonne de la force à ces promesses biotechnologiques, dont l’emblème fut à une époque les organismes génétiquement modifiés (OGM) en agriculture. L’hyperséquençage servirait de soutien à de nouvelles approches d’adaptation ou de conservation ex-situ : par exemple migration d’individus de populations végétales « assistée » par l’information génétique qu’ils portent, voire évolution dirigée des populations à l’aide de technologies de modification de l’ADN de type CRISPR-Cas917. Ces projets techno-fix18 révèlent une posture anthropocentrée d’assistance aux écosystèmes, abîmés par nos propres activités, à l’aide de l’hyperséquençage et des biotechnologies.
Martin Buber, il y a cent ans, évoquant un arbre, qui pourrait symboliser tout être vivant, tout sujet d’étude, écrivait : « Je peux le volatiliser et l’éterniser en le réduisant à un nombre, à un pur rapport numéral19 ». Ainsi, le chiffrage des vivants, obtenu avec des données de plus en plus nombreuses, émises « depuis nulle part », est-il en mesure d’offrir un déchiffrage des raisons du déclin de la biodiversité ? Ou plutôt une copie abstraite d’un monde construit sur des nombres20 ?
La biodiversité sur l’autel du productivisme moléculaire ?
La multiplication des données moléculaires peut être mise en regard de la multiplication des publications faisant état de l’effondrement de la biodiversité. Le nombre d’espèces inscrites sur la liste rouge de l’IUCN, qui a quadruplé en 25 ans, illustre cet effondrement ; de 10 533 en 1996-1998 à 41 459 en 2022. En 2019, la plateforme intergouvernementale scientifique et politique sur la biodiversité et les services écosystémiques (IPBES) estimait « qu’environ 1 million d’espèces animales et végétales sont aujourd’hui menacées d’extinction, notamment au cours des prochaines décennies, ce qui n’a jamais eu lieu auparavant dans l’histoire de l’humanité ». Les populations de plantes et d’animaux, sujets de nos recherches depuis des décennies, sont maintenant en danger de disparition dans leur environnement. Le fait que l’intensité de la crise croisse malgré la prolifération des données ne signale pas a priori que nos recherches soient inutiles, voire dommageables. D’ailleurs les causes de l’extinction de la biodiversité sont connues : pesticides, déforestation, surexploitation des ressources, dégradation des habitats, économie de croissance carbonée, etc.
Notre communauté n’échappe ainsi sans doute pas à la confusion entre progrès et renouvellement incessant des objets et des techniques.
Cependant, un regard rétrospectif révèle que nos disciplines participent à l’intensification de la pression sur les écosystèmes. Elles font en effet appel à de la puissance technique en constante augmentation, consommatrice de matériaux et d’énergie. Ces techniques se sont de plus « démocratisées » : d’abord réservées à des laboratoires de génétique particulièrement équipés, elles deviennent de plus en plus accessibles, ce phénomène étant généralement considéré comme un « progrès » — dont le sens mériterait d’être questionné. Notre communauté n’échappe ainsi sans doute pas à la confusion entre progrès et renouvellement incessant des objets et des techniques. Nous n’échappons pas non plus au productivisme et à l’hyperconsumérisme ambiant, alimentés par l’industrie qui produit, entre autres, les machines de séquençage et d’analyse de données.
Le tour technique pris par les études de génétique des populations constitue une des illustrations des pressions croissantes des technologies humaines sur l’environnement. Ces pressions mettent à mal les milieux et les vivants qui les occupent. Là est la contradiction actuelle de notre discipline : nos méthodes, dont le but premier est d’étudier la biodiversité, portent en elles-mêmes une partie des causes de l’aggravation de l’effondrement de cette même biodiversité. La quantification exacte de l’empreinte écologique des activités des laboratoires de recherche est « un champ émergent de la littérature » porté en France par le GDR Labos 1point5 21. Cependant la massification de l’acquisition de données repose sur une utilisation accrue de machines diverses et variées pour stocker les échantillons, extraire leurs ADN, les séquencer. L’analyse des grandes quantités de données ainsi produites demande des calculs très nombreux et repose de ce fait sur une utilisation accrue de fermes de serveurs informatiques. Enfin, très souvent, l’échantillonnage d’une espèce à l’échelle (inter)-continentale, se fait au prix de nombreux transports aériens, sans compter, à l’issue de ces travaux, l’empreinte écologique liée à la présentation des résultats dans des colloques internationaux 22.

Peut-on alors souhaiter que les seules traces qui nous resteront des espèces que nous étudions soient des extraits d’ADN dans nos « ultracongélateurs » à -80°C, consommateurs d’énergie, ou des génomes séquencés sauvegardés dans des serveurs, également énergivores, à la localisation incertaine, et peut-être lointaine ?
Plaidoyer pour une génétique des populations vivante
Nous nous sommes intéressées ici, en génétique des populations, à une forme d’obsolescence des techniques de marquage utilisées pour étudier la diversité intraspécifique, au cours des 30 dernières années. Les techniques de séquençage, qui ont pris la place de la plupart des techniques précédentes, s’accompagnent d’une accumulation de données qui sont réputées améliorer nos connaissances du vivant.
Dans un contexte de financement de la recherche principalement sur projets, cette accumulation croissante de données est cependant basée d’une part sur les promesses des scientifiques, dans leurs réponses aux appels d’offre, de contribuer à résoudre la crise de la biodiversité actuelle et d’autre part sur le technosolutionnisme assumé des politiques, qui font régulièrement référence à la recherche, comme source de solutions face aux crises climatiques et écologiques.
La question de l’empreinte écologique des techniques déployées pour les études sur le vivant gagnerait à être examinée. Certes, les empreintes d’autres activités humaines polluantes, y compris dans la recherche, par exemple aérospatiale, pourraient être jugées plus préoccupantes que la nôtre. Cependant, se cacher derrière ces dernières, en minimisant et en négligeant la trace environnementale de nos métiers, revient à participer aux discours qui retardent l’action climatique. Ceux-là même qui empêchent la mise en œuvre d’actions pour protéger l’environnement et les vivants qui l’habitent (une attitude qualifiée d’« aquoibonisme » ou, en anglais, de « whataboutism23 »).
Nos démarches et nos outils doivent alors être interrogés sur leur caractère potentiellement non convivial : hétéronomes et dans la démesure24. Leur statut de zombification, de déchets, doit également être questionné. Les « technologies zombies » sont en effet basées sur des ressources finies et ne parviennent pas à mourir ou à disparaître, restant sur terre sous forme de déchets ; au contraire des « technologies vivantes25 ».
Si ni nos institutions, ni une forme de débat « citoyen » ne lancent la réflexion sur ces questions, notre communauté doit-elle continuer néanmoins à accumuler tête baissée ces fameuses données, ou bien doit-elle s’organiser pour faire évoluer ses activités en conséquence ? Quels sens pouvons-nous redonner à notre discipline ?
À quoi pourrait ressembler une génétique des populations plus sobre, plus conviviale, plus vivante ?
Nous plaidons ainsi pour une génétique des populations d’abord soucieuse de ses questions de recherche et de leur intérêt, avant de l’être de ses outils. Une recherche vivante et conviviale, mais aussi plus sobre en technologie, afin de diminuer nos empreintes, de revenir à l’essence de la recherche, de retrouver du sens et ne pas contribuer, par notre accumulation d’expérimentations et de données, à la « grande accélération » : cette mise en évidence du parallèle des croissances exponentielles, depuis 1950, entre les données concernant les activités humaines et des données relatives à la biogéosphère26.
À quoi pourrait ressembler une génétique des populations plus sobre, plus conviviale, plus vivante ? Une première voie, qui ne propose pas de « couper les brins d’ADN à la serpe » — comme nous l’a suggéré un relecteur — serait de sortir de l’hubris, de la démesure, en terme de quantité de données produites et analysées et de mettre fin à l’exhaustivité aveugle que recèle l’approche du « tout séquençage ». Une seconde voie serait aussi dans un vrai partage des données produites car la plupart du temps, ces données peuvent permettre de répondre à quantité de questions ; une approche plus conviviale pourrait alors permettre de mettre fin à l’accumulation de données, directement reliée à la concentration d’argent dans quelques grands laboratoires, et de renouer avec des formes collaboratives de recherche où un jeu de données de séquençage est réellement discuté et partagé entre plusieurs scientifiques. Une génétique des populations plus vivante, enfin, renouerait avec les pratiques d’observation expérimentales, à l’extérieur des laboratoires et en dehors des machines. Il s’agirait là d’une démarche qui irait, par exemple, à rebours de l’observation des espèces par drone, mais qui redonnerait sa place à l’observateur humain, en contact direct avec le terrain, sans intermédiaire ultra-technique, qui mettent à distance notre relation aux vivants.

Ainsi, dans notre pratique de chercheuses, nous nous sommes parfois interrogées, en rencontrant des démarches de recherche contrastées, si une question scientifique bien posée, associée à de petites expériences bien conçues pour y répondre n’était finalement pas plus souhaitable que de gros projets aux questions parfois complexes et floues, avec des analyses sur de nombreux individus localisés sur de grandes surfaces, aux résultats parfois également complexes et flous. Avec cette croyance associée que de grands jeux de données, du fait de leur taille, permettraient la découverte d’un message qui s’y trouverait caché. Les mégadonnées (big data), dont la présence s’est accrue dans nos disciplines ces dernières décennies, sont associées à de nouvelles façons de travailler avec les données : si elles posent des questions et des difficultés sur la façon de les partager, elles ne correspondent pas forcément à de nouvelles façon de faire de la recherche27. De plus, ces mégadonnées peuvent se trouver sujettes à une forme de fétichisme28.
Notons que ces questionnements, touchant spécifiquement la pratique de la génétique des populations, peuvent s’appliquer à d’autres activités scientifiques et prendre alors un tour plus général. Plus globalement, nous relions notre questionnement sur notre discipline aux appels des scientifiques critiques de la technologie des années 197029, aux mouvements de slow science30, mais également, et enfin, à une repolitisation explicite de notre discipline telle qu’elle est proposée par des philosophes31 et par les ateliers d’écologie politique 32.
Face au constat de notre impuissance politique à mettre en œuvre efficacement la conservation des écosystèmes, faut-il voir l’hypersurveillance (sous forme d’accumulation de données et d’indicateurs) dont nous les entourons comme une diversion à cette impuissance ? Tout en nous aveuglant sur le fait que le technolutionnisme contribue à détruire les environnements, nécessaires au maintien de la biodiversité.
À l’inverse, Bernadette Bensaude-Vincent, philosophe des sciences, nous suggère une réflexion temporelle que nous pourrions appliquer à nos approches moléculaires. Il s’agirait d’inventer un temps-paysage :
« Contrairement à la flèche du temps [le temps ne s’écoule que dans une seule direction], qui refuse la diversité en alignant des techniques différentes sur une séquence chronologique, le temps-paysage oblige à penser un monde en devenir où une multitude de techniques diverses et variées, anciennes ou nouvelles, inscrivent leur histoire en croisant leurs devenirs. Dans cette perspective, la question de la durabilité des modes de production et de consommation ne se pose plus en termes de croissance ou d’effondrement (en regardant vers l’avenir) ; elle se centre sur le présent pour considérer comment nos choix technologiques interfèrent avec d’autres temporalités. En faisant un effort d’imagination pour composer avec elles des temps-paysages durables, on peut espérer laisser en héritage autre chose qu’un mémorial de toxicité. »
Bernadette Bensaude-Vincent, 2021. Temps-Paysage. Pour une écologie des crises, Le Pommier, p. 19.
À lire sur Terrestres à propos du livre Temps-Paysage : Marine Fauché, « Orchestrer sans métronome », décembre 2021
Remerciements
L’idée de ce billet est née lors du séminaire « Vivants et technologies » que nous avons organisé pour l’UMR Biogeco le 24 juin 2022. Nous remercions notamment Philippe Bihouix d’avoir débattu avec nous à cette occasion.
Nous remercions Nicolas Berger, Antoine Bouzin, Cyril Dutech, Felix Garnier, Jérôme Santolini ainsi que les relecteur·ices anonymes de la revue Terrestres.
Crédit de l’illustration de Une – Karolina Kołodziejczak
Notes
- Acide désoxyribonucléique, présent dans les chromosomes, support de l’information génétique.[↩]
- Mendel G., 1866. Versuche über Pflanzenhybriden. Verhandlungen des naturforschenden Vereines in Brno, Bd. IV für das Jahr 1865, Abhandlungen, 3–47.[↩]
- Un type de marqueur génétique pour lequel la variabilité de l’endroit du génome que l’on analyse est révélée directement.[↩]
- Détermination de l’ordonnancement linéaire des nucléotides, bases qui constituent la séquence d’une molécule d’ADN ou d’un génome complet.[↩]
- Ensemble du matériel génétique d’une espèce.[↩]
- En anglais, single nucleotide polymorphism. Le polymorphisme nucléotidique — c’est-à-dire la propriété des individus d’une espèce de présenter plusieurs formes différentes — correspond à la variation d’une seule base du génome entre deux fragments d’ADN. L’utilisation de ces marqueurs génétiques permet de quantifier les différences entre individus, et entre populations d’une même espèce.[↩]
- Gerber S., Mariette S., Streiff R., Bodénès C., Kremer A. 2000. « Comparison of microsatellites and AFLP markers for parentage analysis ». Molecular Ecology 9:1037-1048 ; Mariette, S., Le Corre, V., Austerlitz, F., Kremer, A. 2002. « Sampling within the genome for measuring within-population diversity: trade-offs between markers ». Molecular Ecology, 11, 1145-1156[↩]
- Matarasso, M. 1963. « Packard Vance, L’art du gaspillage ». Revue française de sociologie, 4(1), pp. 85-87. www.persee.fr/doc/rfsoc_0035-2969_1963_num_4_1_7132[↩]
- Molecular Ecology, 2023. Overview. Aims and Scope.
https://onlinelibrary.wiley.com/page/journal/1365294x/homepage/productinformation.html[↩] - Bensaude-Vincent, B. 2021. Temps-Paysage. Pour une écologie des crises. Le Pommier, p35[↩]
- Joly, P.B. 2010. ‘On the Economics of Techno-scientific Promises’, in Akrich, M., Barthe, Y., Muniesa, F., Mustar, P. (eds.) Débordements. Mélanges offerts à Michel Callon, Paris : Presse des Mines, pp.203-222 ; Joly, P.B. 2013. « À propos de l’économie des promesses techno-scientifiques ». Futuris 2013 : La recherche et l’innovation en France, Odile Jacob ; Compagnon, D., Saint-Martin, A. 2019. « La technique : promesse, mirage et fatalité », Socio. La nouvelle revue des sciences sociales 12, 7–25[↩]
- Huesemann, M., Huesemann, J. 2011. Techno-fix: why technology won’t save us or the environment. Gabriola Island, B.C: New Society Publishers.[↩]
- Segal, H.P. 2017. « Practical Utopias: America as Techno-Fix Nation ». Utopian Studies, 28(2), 231–246[↩]
- Cook, J. 2016. « Young adults hopes for the long-term future: from re-enchantment with technology to faith in humanity », Journal of Youth Studies, 19(4), pp. 517–532 ; Devictor, V. 2019. « Anthropocène, dis-moi, combien tu t’appelles ? » Terrestres. https://www.terrestres.org/2019/05/26/anthropocene-dis-moi-combien-tu-tappelles/[↩]
- Discipline scientifique qui a pour objet d’inventorier l’ensemble des gènes d’un organisme vivant et d’en étudier les fonctions[↩]
- ANR 2023. Recherche projet “ génétique ”, “ population ” : https://anr.fr/fr/projets-finances-et-impact/[↩]
- Système de biologie moléculaire qui permet de cibler une séquence particulière d’ADN et d’y effectuer une cassure, pour ensuite y apporter des modifications.[↩]
- De technology et fix, réparer : solution technologique à un problème.[↩]
- Buber, M. 1923. Je et tu. Éditions Aubier, 2012, p 39-41[↩]
- Devictor 2019, op. cit.[↩]
- Ben Ari, T. et Berné, O. 2023. « La frugalité, un terme un peu radioactif », Socio. La nouvelle revue des sciences sociales 17, 67-82.[↩]
- Berné, O., Agier, L., Hardy, A., Lellouch, E., Aumont, O., Mariette, J., Ben-Ari, T. 2022. « The carbon footprint of scientific visibility », Environmental Research Letters, 17, 124008.[↩]
- Lamb, W., Mattioli, G., Levi, S., Roberts, J., Capstick, S., Creutzig, F., Minx J.C., Müller-Hansen F., Culhane T., Steinberger, J. 2020. “Discourses of climate delay”. Global Sustainability, 3, E17.[↩]
- Illich, I. 1973. La convivialité. Paris, Seuil[↩]
- Monnin A. 2020 « Au-delà du low tech : technologies zombies, soutenabilité et inventions. Interview croisée de José Halloy et Nicolas Nova ». Low tech: face au tout-numérique, se réapproprier les technologies. Passerelle n° 21. pp.120-128, Éditions Ritimo[↩]
- Crutzen, P., Steffen, W. 2003. « How long have we been in the Anthropocene era? » Climatic Change 61, 251-257, cité par Bensaude-Vincent, 2021, figure 2, p. 36[↩]
- Leonelli, S. 2014. « What difference does quantity make? On the epistemology of Big Data in biology », Big Data & Society, 1(1), p. 205395171453439. https://doi.org/10.1177/2053951714534395.[↩]
- D’Ignazio, C. Klein, L. 2020. « The Numbers Don’t Speak for Themselves ». in Data Feminism. Cambridge MA, MIT Press.
https://data-feminism.mitpress.mit.edu/pub/czq9dfs5/release/3[↩] - Jarrige, F. 2017. « Technocritique et écologie (années 1970) », La pensée écologique 1. https://lapenseeecologique.com/jarrige-francois-techno-critique-et-ecologie-annees-1970/[↩]
- Stengers, I., William, J. 2013. Une Autre Science Est Possible ! : Manifeste Pour Un Ralentissement Des Sciences. Paris, La Découverte[↩]
- Stengers, I., 2023. « Lettre aux scientifiques qui lisent les rapports du GIEC ». Socialter. https://www.socialter.fr/article/isabelle-stengers-lettre-aux-scientifiques-qui-lisent-les-rapports-du-giec[↩]
- Atecopol, Manifeste, 2018. https://atecopol.hypotheses.org/manifeste-de-latelier-decologie-politique-toulousain[↩]